Prediksi Diagnosis Kanker Payudara#
Pendahuluan#
Kanker payudara merupakan salah satu jenis kanker yang paling umum dan menjadi penyebab utama kematian pada wanita di seluruh dunia. Menurut data dari World Health Organization (WHO), deteksi dini terhadap kanker payudara sangat penting untuk meningkatkan peluang kesembuhan dan menurunkan tingkat kematian. Salah satu metode yang dapat digunakan untuk mendeteksi kanker payudara adalah dengan menganalisis karakteristik sel kanker menggunakan teknologi komputer.
Perkembangan teknologi di bidang kecerdasan buatan, khususnya machine learning, memungkinkan sistem komputer untuk melakukan analisis data secara otomatis dan menghasilkan prediksi yang akurat. Dengan memanfaatkan dataset medis yang telah tersedia, seperti Breast Cancer Wisconsin (Diagnostic) Dataset. Algoritma machine learning dapat dilatih untuk membedakan antara tumor jinak (benign) dan ganas (malignant).
Data Understanding#
Sumber Data#
Dataset dapat dilihat pada link berikut ini :
https://archive.ics.uci.edu/dataset/17/breast+cancer+wisconsin+diagnostic
Dataset Breast Cancer Wisconsin (Diagnostic) dari UCI berisi data medis yang digunakan untuk memprediksi apakah suatu tumor bersifat ganas (malignant) atau jinak (benign) berdasarkan hasil pemeriksaan mikroskopik sel kanker.
Tujuan#
Dataset ini digunakan untuk membangun model klasifikasi untuk memprediksi ganas atau jinak nya tumor kanker payudara pada pasien, guna mendukung penelitian dan diagnosis medis.
Integrasi Data#
Untuk mengolah data dari UCI Dataset, maka perlu menginstal pustaka (package) yang diperlukan. Instalasi ini bertujuan agar data yang tersedia di UCI dapat diunduh secara langsung ke dalam lingkungan Python untuk dianalisis lebih lanjut. Langkah pengambilan data dapat dilakukan dengan mengikuti instruksi yang muncul setelah menekan tombol “Import in Python” pada halaman dataset yang dipilih. Perintah tersebut akan memberikan kode Python yang siap digunakan untuk mengunduh dan memuat dataset secara otomatis.
pip install ucimlrepo
Requirement already satisfied: ucimlrepo in /home/codespace/.python/current/lib/python3.12/site-packages (0.0.7)
Requirement already satisfied: pandas>=1.0.0 in /home/codespace/.local/lib/python3.12/site-packages (from ucimlrepo) (2.2.3)
Requirement already satisfied: certifi>=2020.12.5 in /home/codespace/.local/lib/python3.12/site-packages (from ucimlrepo) (2024.8.30)
Requirement already satisfied: numpy>=1.26.0 in /home/codespace/.python/current/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (1.26.4)
Requirement already satisfied: python-dateutil>=2.8.2 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2.9.0.post0)
Requirement already satisfied: pytz>=2020.1 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2024.2)
Requirement already satisfied: tzdata>=2022.7 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.0.0->ucimlrepo) (2024.2)
Requirement already satisfied: six>=1.5 in /home/codespace/.local/lib/python3.12/site-packages (from python-dateutil>=2.8.2->pandas>=1.0.0->ucimlrepo) (1.17.0)
Note: you may need to restart the kernel to use updated packages.
from ucimlrepo import fetch_ucirepo
import pandas as pd
# fetch dataset
breast_cancer_wisconsin_diagnostic = fetch_ucirepo(id=17)
# data (as pandas dataframes)
X = breast_cancer_wisconsin_diagnostic.data.features
y = breast_cancer_wisconsin_diagnostic.data.targets
# gabungkan fitur dan target jadi satu dataframe
df = pd.concat([X, y], axis=1)
# simpan ke file CSV
df.to_csv("breast.csv", index=False)
# metadata
print(breast_cancer_wisconsin_diagnostic.metadata)
# variable information
print(breast_cancer_wisconsin_diagnostic.variables)
{'uci_id': 17, 'name': 'Breast Cancer Wisconsin (Diagnostic)', 'repository_url': 'https://archive.ics.uci.edu/dataset/17/breast+cancer+wisconsin+diagnostic', 'data_url': 'https://archive.ics.uci.edu/static/public/17/data.csv', 'abstract': 'Diagnostic Wisconsin Breast Cancer Database.', 'area': 'Health and Medicine', 'tasks': ['Classification'], 'characteristics': ['Multivariate'], 'num_instances': 569, 'num_features': 30, 'feature_types': ['Real'], 'demographics': [], 'target_col': ['Diagnosis'], 'index_col': ['ID'], 'has_missing_values': 'no', 'missing_values_symbol': None, 'year_of_dataset_creation': 1993, 'last_updated': 'Fri Nov 03 2023', 'dataset_doi': '10.24432/C5DW2B', 'creators': ['William Wolberg', 'Olvi Mangasarian', 'Nick Street', 'W. Street'], 'intro_paper': {'ID': 230, 'type': 'NATIVE', 'title': 'Nuclear feature extraction for breast tumor diagnosis', 'authors': 'W. Street, W. Wolberg, O. Mangasarian', 'venue': 'Electronic imaging', 'year': 1993, 'journal': None, 'DOI': '10.1117/12.148698', 'URL': 'https://www.semanticscholar.org/paper/53f0fbb425bc14468eb3bf96b2e1d41ba8087f36', 'sha': None, 'corpus': None, 'arxiv': None, 'mag': None, 'acl': None, 'pmid': None, 'pmcid': None}, 'additional_info': {'summary': 'Features are computed from a digitized image of a fine needle aspirate (FNA) of a breast mass. They describe characteristics of the cell nuclei present in the image. A few of the images can be found at http://www.cs.wisc.edu/~street/images/\r\n\r\nSeparating plane described above was obtained using Multisurface Method-Tree (MSM-T) [K. P. Bennett, "Decision Tree Construction Via Linear Programming." Proceedings of the 4th Midwest Artificial Intelligence and Cognitive Science Society, pp. 97-101, 1992], a classification method which uses linear programming to construct a decision tree. Relevant features were selected using an exhaustive search in the space of 1-4 features and 1-3 separating planes.\r\n\r\nThe actual linear program used to obtain the separating plane in the 3-dimensional space is that described in: [K. P. Bennett and O. L. Mangasarian: "Robust Linear Programming Discrimination of Two Linearly Inseparable Sets", Optimization Methods and Software 1, 1992, 23-34].\r\n\r\nThis database is also available through the UW CS ftp server:\r\nftp ftp.cs.wisc.edu\r\ncd math-prog/cpo-dataset/machine-learn/WDBC/', 'purpose': None, 'funded_by': None, 'instances_represent': None, 'recommended_data_splits': None, 'sensitive_data': None, 'preprocessing_description': None, 'variable_info': '1) ID number\r\n2) Diagnosis (M = malignant, B = benign)\r\n3-32)\r\n\r\nTen real-valued features are computed for each cell nucleus:\r\n\r\n\ta) radius (mean of distances from center to points on the perimeter)\r\n\tb) texture (standard deviation of gray-scale values)\r\n\tc) perimeter\r\n\td) area\r\n\te) smoothness (local variation in radius lengths)\r\n\tf) compactness (perimeter^2 / area - 1.0)\r\n\tg) concavity (severity of concave portions of the contour)\r\n\th) concave points (number of concave portions of the contour)\r\n\ti) symmetry \r\n\tj) fractal dimension ("coastline approximation" - 1)', 'citation': None}}
name role type demographic description units \
0 ID ID Categorical None None None
1 Diagnosis Target Categorical None None None
2 radius1 Feature Continuous None None None
3 texture1 Feature Continuous None None None
4 perimeter1 Feature Continuous None None None
5 area1 Feature Continuous None None None
6 smoothness1 Feature Continuous None None None
7 compactness1 Feature Continuous None None None
8 concavity1 Feature Continuous None None None
9 concave_points1 Feature Continuous None None None
10 symmetry1 Feature Continuous None None None
11 fractal_dimension1 Feature Continuous None None None
12 radius2 Feature Continuous None None None
13 texture2 Feature Continuous None None None
14 perimeter2 Feature Continuous None None None
15 area2 Feature Continuous None None None
16 smoothness2 Feature Continuous None None None
17 compactness2 Feature Continuous None None None
18 concavity2 Feature Continuous None None None
19 concave_points2 Feature Continuous None None None
20 symmetry2 Feature Continuous None None None
21 fractal_dimension2 Feature Continuous None None None
22 radius3 Feature Continuous None None None
23 texture3 Feature Continuous None None None
24 perimeter3 Feature Continuous None None None
25 area3 Feature Continuous None None None
26 smoothness3 Feature Continuous None None None
27 compactness3 Feature Continuous None None None
28 concavity3 Feature Continuous None None None
29 concave_points3 Feature Continuous None None None
30 symmetry3 Feature Continuous None None None
31 fractal_dimension3 Feature Continuous None None None
missing_values
0 no
1 no
2 no
3 no
4 no
5 no
6 no
7 no
8 no
9 no
10 no
11 no
12 no
13 no
14 no
15 no
16 no
17 no
18 no
19 no
20 no
21 no
22 no
23 no
24 no
25 no
26 no
27 no
28 no
29 no
30 no
31 no
Eksplorasi Data#
Menampilkan 5 data dari dataset
display(df)
| radius1 | texture1 | perimeter1 | area1 | smoothness1 | compactness1 | concavity1 | concave_points1 | symmetry1 | fractal_dimension1 | ... | texture3 | perimeter3 | area3 | smoothness3 | compactness3 | concavity3 | concave_points3 | symmetry3 | fractal_dimension3 | Diagnosis | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 17.99 | 10.38 | 122.80 | 1001.0 | 0.11840 | 0.27760 | 0.30010 | 0.14710 | 0.2419 | 0.07871 | ... | 17.33 | 184.60 | 2019.0 | 0.16220 | 0.66560 | 0.7119 | 0.2654 | 0.4601 | 0.11890 | M |
| 1 | 20.57 | 17.77 | 132.90 | 1326.0 | 0.08474 | 0.07864 | 0.08690 | 0.07017 | 0.1812 | 0.05667 | ... | 23.41 | 158.80 | 1956.0 | 0.12380 | 0.18660 | 0.2416 | 0.1860 | 0.2750 | 0.08902 | M |
| 2 | 19.69 | 21.25 | 130.00 | 1203.0 | 0.10960 | 0.15990 | 0.19740 | 0.12790 | 0.2069 | 0.05999 | ... | 25.53 | 152.50 | 1709.0 | 0.14440 | 0.42450 | 0.4504 | 0.2430 | 0.3613 | 0.08758 | M |
| 3 | 11.42 | 20.38 | 77.58 | 386.1 | 0.14250 | 0.28390 | 0.24140 | 0.10520 | 0.2597 | 0.09744 | ... | 26.50 | 98.87 | 567.7 | 0.20980 | 0.86630 | 0.6869 | 0.2575 | 0.6638 | 0.17300 | M |
| 4 | 20.29 | 14.34 | 135.10 | 1297.0 | 0.10030 | 0.13280 | 0.19800 | 0.10430 | 0.1809 | 0.05883 | ... | 16.67 | 152.20 | 1575.0 | 0.13740 | 0.20500 | 0.4000 | 0.1625 | 0.2364 | 0.07678 | M |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 564 | 21.56 | 22.39 | 142.00 | 1479.0 | 0.11100 | 0.11590 | 0.24390 | 0.13890 | 0.1726 | 0.05623 | ... | 26.40 | 166.10 | 2027.0 | 0.14100 | 0.21130 | 0.4107 | 0.2216 | 0.2060 | 0.07115 | M |
| 565 | 20.13 | 28.25 | 131.20 | 1261.0 | 0.09780 | 0.10340 | 0.14400 | 0.09791 | 0.1752 | 0.05533 | ... | 38.25 | 155.00 | 1731.0 | 0.11660 | 0.19220 | 0.3215 | 0.1628 | 0.2572 | 0.06637 | M |
| 566 | 16.60 | 28.08 | 108.30 | 858.1 | 0.08455 | 0.10230 | 0.09251 | 0.05302 | 0.1590 | 0.05648 | ... | 34.12 | 126.70 | 1124.0 | 0.11390 | 0.30940 | 0.3403 | 0.1418 | 0.2218 | 0.07820 | M |
| 567 | 20.60 | 29.33 | 140.10 | 1265.0 | 0.11780 | 0.27700 | 0.35140 | 0.15200 | 0.2397 | 0.07016 | ... | 39.42 | 184.60 | 1821.0 | 0.16500 | 0.86810 | 0.9387 | 0.2650 | 0.4087 | 0.12400 | M |
| 568 | 7.76 | 24.54 | 47.92 | 181.0 | 0.05263 | 0.04362 | 0.00000 | 0.00000 | 0.1587 | 0.05884 | ... | 30.37 | 59.16 | 268.6 | 0.08996 | 0.06444 | 0.0000 | 0.0000 | 0.2871 | 0.07039 | B |
569 rows × 31 columns
Visualisasi distribusi data
pip install matplotlib seaborn
Requirement already satisfied: matplotlib in /home/codespace/.python/current/lib/python3.12/site-packages (3.10.3)
Requirement already satisfied: seaborn in /home/codespace/.local/lib/python3.12/site-packages (0.13.2)
Requirement already satisfied: contourpy>=1.0.1 in /home/codespace/.local/lib/python3.12/site-packages (from matplotlib) (1.3.1)
Requirement already satisfied: cycler>=0.10 in /home/codespace/.local/lib/python3.12/site-packages (from matplotlib) (0.12.1)
Requirement already satisfied: fonttools>=4.22.0 in /home/codespace/.local/lib/python3.12/site-packages (from matplotlib) (4.55.3)
Requirement already satisfied: kiwisolver>=1.3.1 in /home/codespace/.local/lib/python3.12/site-packages (from matplotlib) (1.4.7)
Requirement already satisfied: numpy>=1.23 in /home/codespace/.python/current/lib/python3.12/site-packages (from matplotlib) (1.26.4)
Requirement already satisfied: packaging>=20.0 in /home/codespace/.local/lib/python3.12/site-packages (from matplotlib) (24.2)
Requirement already satisfied: pillow>=8 in /home/codespace/.local/lib/python3.12/site-packages (from matplotlib) (11.0.0)
Requirement already satisfied: pyparsing>=2.3.1 in /home/codespace/.local/lib/python3.12/site-packages (from matplotlib) (3.2.0)
Requirement already satisfied: python-dateutil>=2.7 in /home/codespace/.local/lib/python3.12/site-packages (from matplotlib) (2.9.0.post0)
Requirement already satisfied: pandas>=1.2 in /home/codespace/.local/lib/python3.12/site-packages (from seaborn) (2.2.3)
Requirement already satisfied: pytz>=2020.1 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.2->seaborn) (2024.2)
Requirement already satisfied: tzdata>=2022.7 in /home/codespace/.local/lib/python3.12/site-packages (from pandas>=1.2->seaborn) (2024.2)
Requirement already satisfied: six>=1.5 in /home/codespace/.local/lib/python3.12/site-packages (from python-dateutil>=2.7->matplotlib) (1.17.0)
Note: you may need to restart the kernel to use updated packages.
import matplotlib.pyplot as plt
import seaborn as sns
# Pastikan kolom Diagnosis ada
kolom_target = 'Diagnosis'
# 1. Visualisasi distribusi diagnosis
plt.figure(figsize=(6, 4))
sns.countplot(data=df, x=kolom_target, hue=kolom_target, palette='Set2', legend=False)
plt.title("Distribusi Diagnosis (Benign vs Malignant)")
plt.xlabel("Diagnosis")
plt.ylabel("Jumlah")
plt.show()
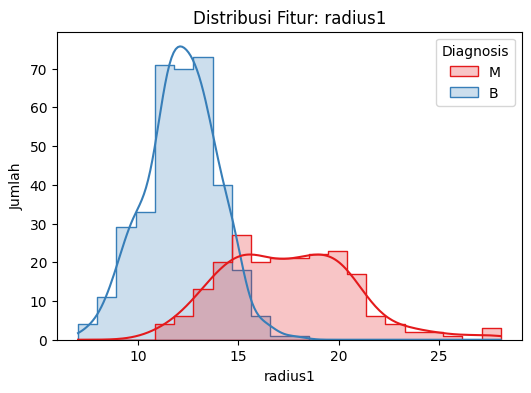
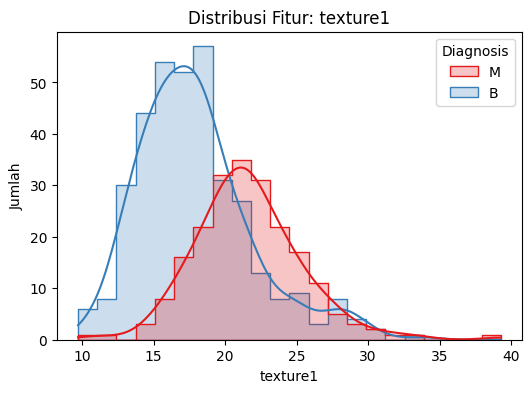
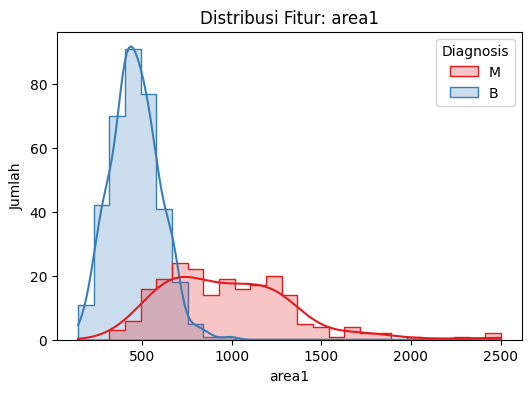
# 2. Visualisasi distribusi fitur (contoh: mean features)
fitur_pilihan = ['radius1', 'texture1', 'area1'] # 1 = mean
for fitur in fitur_pilihan:
plt.figure(figsize=(6, 4))
sns.histplot(data=df, x=fitur, hue=kolom_target, kde=True, palette='Set1', element='step')
plt.title(f"Distribusi Fitur: {fitur}")
plt.xlabel(fitur)
plt.ylabel("Jumlah")
plt.show()
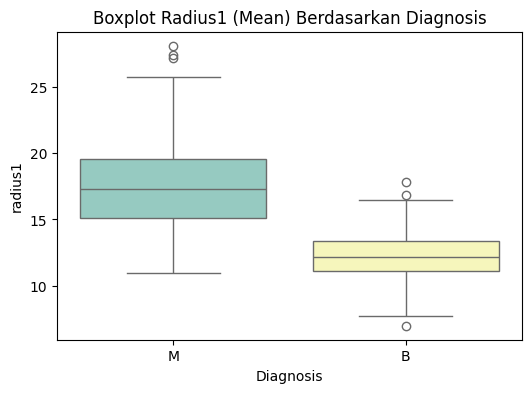
# 3. Boxplot berdasarkan diagnosis
plt.figure(figsize=(6, 4))
sns.boxplot(data=df, x=kolom_target, y='radius1', palette='Set3')
plt.title("Boxplot Radius1 (Mean) Berdasarkan Diagnosis")
plt.xlabel("Diagnosis")
plt.ylabel("radius1")
plt.show()
/tmp/ipykernel_91439/566655522.py:28: FutureWarning:
Passing `palette` without assigning `hue` is deprecated and will be removed in v0.14.0. Assign the `x` variable to `hue` and set `legend=False` for the same effect.
sns.boxplot(data=df, x=kolom_target, y='radius1', palette='Set3')
Struktur Dataset#
Fitur dan Label#
df.shape
(569, 31)
Terdapat 569 baris data dan 31 kolom
df.columns
Index(['radius1', 'texture1', 'perimeter1', 'area1', 'smoothness1',
'compactness1', 'concavity1', 'concave_points1', 'symmetry1',
'fractal_dimension1', 'radius2', 'texture2', 'perimeter2', 'area2',
'smoothness2', 'compactness2', 'concavity2', 'concave_points2',
'symmetry2', 'fractal_dimension2', 'radius3', 'texture3', 'perimeter3',
'area3', 'smoothness3', 'compactness3', 'concavity3', 'concave_points3',
'symmetry3', 'fractal_dimension3', 'Diagnosis'],
dtype='object')
Fitur adalah variabel atau atribut yang digunakan sebagai input dalam proses analisis data dan pemodelan machine learning. Fitur mewakili karakteristik, sifat, atau informasi yang dimiliki oleh suatu data. Terdapat 30 fitur dan 1 class pada dataset ini yaitu :
Fitur Mean (akhiran 1)
radius1: Rata-rata jarak dari pusat ke batas inti sel.
texture1: Rata-rata variasi tingkat keabuan (tekstur permukaan inti sel).
perimeter1: Rata-rata keliling dari inti sel.
area1: Rata-rata luas area inti sel.
smoothness1: Rata-rata kelicinan batas inti sel.
compactness1: Rata-rata kekompakan inti sel (perimeter² / area - 1.0).
concavity1: Rata-rata tingkat cekungan pada kontur inti sel.
concave_points1: Rata-rata jumlah titik-titik cekung pada kontur inti sel.
symmetry1: Rata-rata tingkat kesimetrian inti sel.
fractal_dimension1: Rata-rata kompleksitas kontur inti sel berdasarkan dimensi fraktal.
Fitur Standard Error (akhiran 2)
radius2: Standard error dari ukuran radius inti sel.
texture2: Standard error dari tekstur inti sel.
perimeter2: Standard error dari keliling inti sel.
area2: Standard error dari luas area inti sel.
smoothness2: Standard error dari kelicinan batas inti sel.
compactness2: Standard error dari kekompakan inti sel.
concavity2: Standard error dari tingkat cekungan kontur inti sel.
concave_points2: Standard error dari jumlah titik cekung pada kontur inti sel.
symmetry2: Standard error dari tingkat simetri inti sel.
fractal_dimension2: Standard error dari kompleksitas kontur inti sel.
Fitur Terburuk (akhiran 3)
radius3: Nilai maksimum radius inti sel (terburuk).
texture3: Nilai maksimum variasi tekstur inti sel.
perimeter3: Nilai maksimum keliling inti sel.
area3: Nilai maksimum luas area inti sel.
smoothness3: Nilai maksimum kelicinan batas inti sel.
compactness3: Nilai maksimum kekompakan inti sel.
concavity3: Nilai maksimum tingkat cekungan kontur inti sel.
concave_points3: Nilai maksimum jumlah titik-titik cekung pada kontur inti sel.
symmetry3: Nilai maksimum tingkat simetri inti sel.
fractal_dimension3: Nilai maksimum kompleksitas kontur inti sel.
Kolom Target
Diagnosis: Label target yang menunjukkan jenis tumor:
M = Malignant (ganas)
B = Benign (jinak)
df.dtypes
radius1 float64
texture1 float64
perimeter1 float64
area1 float64
smoothness1 float64
compactness1 float64
concavity1 float64
concave_points1 float64
symmetry1 float64
fractal_dimension1 float64
radius2 float64
texture2 float64
perimeter2 float64
area2 float64
smoothness2 float64
compactness2 float64
concavity2 float64
concave_points2 float64
symmetry2 float64
fractal_dimension2 float64
radius3 float64
texture3 float64
perimeter3 float64
area3 float64
smoothness3 float64
compactness3 float64
concavity3 float64
concave_points3 float64
symmetry3 float64
fractal_dimension3 float64
Diagnosis object
dtype: object
Semua fitur memiliki tipe data float sedangkan target/class bertipe data object/kategorikal
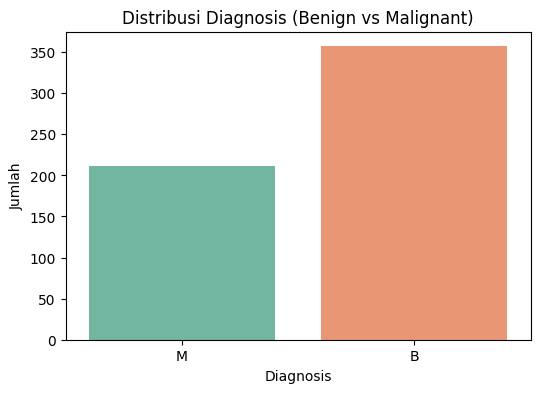
df.groupby('Diagnosis').size()
Diagnosis
B 357
M 212
dtype: int64
Pada kelas Malignant (ganas) sebanyak 212 data, sedangkan kelas Benign (jinak) sebanyak 357 data.
Mengidentifikasi Kualitas Dataset#
Cek Missing Value (Nilai Hilang)
Memeriksa apakah ada data yang kosong (NaN) pada kolom fitur atau target.
Tidak ada mising value pada dataset
print(df.isnull().sum())
radius1 0
texture1 0
perimeter1 0
area1 0
smoothness1 0
compactness1 0
concavity1 0
concave_points1 0
symmetry1 0
fractal_dimension1 0
radius2 0
texture2 0
perimeter2 0
area2 0
smoothness2 0
compactness2 0
concavity2 0
concave_points2 0
symmetry2 0
fractal_dimension2 0
radius3 0
texture3 0
perimeter3 0
area3 0
smoothness3 0
compactness3 0
concavity3 0
concave_points3 0
symmetry3 0
fractal_dimension3 0
Diagnosis 0
dtype: int64
Cek Duplikat
Mengidentifikasi apakah ada baris data yang terduplikasi.
Tidak ada data duplikat pada dataset ini
df.duplicated().sum()
0
Preprocessing Data#
Transformasi Data#
Pada Dataset ini class yang ada di kolom Diagnosis bertipe data kategorikal, maka perlu dilakukan transformasi data yaitu mengubah data kategorikal ke data numerik. Kelas Malignant menjadi 1 dan Benign menjadi 0.
# Ubah nilai Diagnosis dari M/B menjadi 1/0
df['Diagnosis'] = df['Diagnosis'].map({'M': 1, 'B': 0})
# Cek hasil transformasi
print(df['Diagnosis'].value_counts())
df_clean.to_csv("encode.csv", index=False)
Diagnosis
0 357
1 212
Name: count, dtype: int64
---------------------------------------------------------------------------
NameError Traceback (most recent call last)
Cell In[12], line 7
4 # Cek hasil transformasi
5 print(df['Diagnosis'].value_counts())
----> 7 df_clean.to_csv("encode.csv", index=False)
NameError: name 'df_clean' is not defined
Mendeteksi dan Menghapus Outlier dengan LOF#
Konsep Local Outlier Factor
Local Outlier Factor (LOF) adalah sebuah metode deteksi outlier berbasis tetangga terdekat (k-NN) yang mengukur seberapa jauh kepadatan lokal sebuah titik data dibandingkan dengan kepadatan tetangganya.
LOF tidak hanya melihat seberapa jauh suatu titik dari titik lainnya, tetapi juga membandingkan kepadatan lokal di sekitarnya. Jadi, LOF bisa mendeteksi outlier yang “lokal”, yaitu titik-titik yang menyimpang dalam wilayah padat maupun wilayah jarang.
Langkah-langkah LOF
Menentukan jumlah tetangga terdekat (k-nearest neighbors)
Menghitung reachability distance untuk setiap titik.
Menghitung local reachability density (LRD).
Menghitung LOF sebagai rasio LRD titik terhadap LRD tetangganya.
Menentukan apakah titik termasuk outlier berdasarkan skor LOF.
import pandas as pd
from sklearn.neighbors import LocalOutlierFactor
# 1. Load file CSV (yang sudah di-encode Diagnosis)
df = pd.read_csv('encode.csv')
# 3. Pisahkan fitur numerik (tanpa Diagnosis dan ID)
fitur = df.drop(columns=['ID', 'Diagnosis','LOF Score', 'Prediksi'])
# 4. Terapkan LOF
lof = LocalOutlierFactor(n_neighbors=20, contamination=0.05)
pred = lof.fit_predict(fitur) # Hasil: 1 (inlier), -1 (outlier)
# 5. Hapus outlier
df_clean = df[pred == 1].copy()
# 6. Tampilkan info pembersihan
print("Jumlah data sebelum dibersihkan:", df.shape[0])
print("Jumlah data setelah dibersihkan :", df_clean.shape[0])
print("Jumlah outlier yang dihapus :", df.shape[0] - df_clean.shape[0])
# 7. Simpan hasil bersih (jika ingin)
df_clean.to_csv("breast_cancer_tanpa_outlier.csv", index=False)
Jumlah data sebelum dibersihkan: 513
Jumlah data setelah dibersihkan : 487
Jumlah outlier yang dihapus : 26
Modelling Klasifikasi Gaussian Naive Bayes#
Gaussian Naive Bayes merupakan salah satu algoritma klasifikasi dalam pembelajaran mesin yang berdasarkan pada teorema Bayes dengan asumsi bahwa antar fitur tidak saling bergantung (naive assumption) dan data numerik berdistribusi normal (Gaussian distribution).
Algoritma ini sangat cocok digunakan untuk data dengan fitur numerik, seperti dataset Breast Cancer Wisconsin (Diagnostic) yang memiliki fitur hasil pengukuran morfologi sel. Gaussian Naive Bayes menghitung peluang masing-masing kelas berdasarkan distribusi normal dari setiap fitur, kemudian memilih kelas dengan probabilitas tertinggi sebagai prediksi.
Tujuan Modeling#
Tujuan dari penerapan Gaussian Naive Bayes pada dataset ini adalah untuk:
Mengklasifikasikan data menjadi dua kelas, yaitu benign (jinak) dan malignant (ganas).
Mengukur performa model dalam mendeteksi kanker payudara berdasarkan fitur-fitur numerik yang tersedia.
Langkah-langkah Modeling#
Membagi Data (Train-Test Split)
Data dibagi menjadi dua bagian menggunakan metode train_test_split :
80% data untuk pelatihan (training)
20% data untuk pengujian (testing) Pembagian dilakukan secara acak dengan parameter random_state untuk hasil yang konsisten.
Pelatihan Model
Model Gaussian Naive Bayes dibuat menggunakan pustaka sklearn.naive_bayes.GaussianNB, dan dilatih pada data training.Prediksi dan Evaluasi Setelah model dilatih, data testing digunakan untuk melakukan prediksi. Evaluasi dilakukan menggunakan beberapa metrik:
Akurasi: Persentase prediksi yang benar dari total data.
Confusion Matrix: Matriks yang menunjukkan jumlah true positive, false positive, true negative, dan false negative.
Classification Report: Menyajikan metrik precision, recall, dan F1-score untuk tiap kelas.
df.columns
Index(['ID', 'radius1', 'texture1', 'perimeter1', 'area1', 'smoothness1',
'compactness1', 'concavity1', 'concave_points1', 'symmetry1',
'fractal_dimension1', 'radius2', 'texture2', 'perimeter2', 'area2',
'smoothness2', 'compactness2', 'concavity2', 'concave_points2',
'symmetry2', 'fractal_dimension2', 'radius3', 'texture3', 'perimeter3',
'area3', 'smoothness3', 'compactness3', 'concavity3', 'concave_points3',
'symmetry3', 'fractal_dimension3', 'Diagnosis', 'LOF Score',
'Prediksi'],
dtype='object')
from sklearn.model_selection import train_test_split
from sklearn.naive_bayes import GaussianNB
from sklearn.metrics import classification_report, confusion_matrix, accuracy_score
import pandas as pd
# 1. Load data tanpa outlier
df = pd.read_csv('breast_cancer_tanpa_outlier.csv')
# 2. Pisahkan fitur dan target
X = df.drop(columns=['Diagnosis', 'ID', 'LOF Score', 'Prediksi'], errors='ignore')
y = df['Diagnosis']
# 3. Split data training dan testing
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# 4. Model Gaussian Naive Bayes
model = GaussianNB()
model.fit(X_train, y_train)
# 5. Prediksi dan evaluasi
y_pred = model.predict(X_test)
print("Akurasi :", accuracy_score(y_test, y_pred))
print("\nConfusion Matrix:\n", confusion_matrix(y_test, y_pred))
print("\nClassification Report:\n", classification_report(y_test, y_pred))
Akurasi : 0.9795918367346939
Confusion Matrix:
[[65 1]
[ 1 31]]
Classification Report:
precision recall f1-score support
0 0.98 0.98 0.98 66
1 0.97 0.97 0.97 32
accuracy 0.98 98
macro avg 0.98 0.98 0.98 98
weighted avg 0.98 0.98 0.98 98
Modelling Klasifikasi Decision Tree#
Decision Tree (pohon keputusan) adalah salah satu algoritma dalam pembelajaran mesin yang digunakan untuk klasifikasi dan regresi, dengan cara membagi data ke dalam kelompok-kelompok berdasarkan serangkaian aturan keputusan (if-then-else) yang diturunkan dari fitur-fitur input. Model ini berbentuk seperti struktur pohon, di mana:
Node merepresentasikan atribut/fitur,
Cabang (branch) mewakili kondisi atau aturan,
Daun (leaf node) merupakan hasil akhir berupa kelas prediksi.
Tujuan Modeling#
Penerapan Decision Tree pada dataset Breast Cancer Wisconsin (Diagnostic) bertujuan untuk:
Membangun model klasifikasi yang mampu memprediksi apakah suatu sampel merupakan kanker ganas (malignant) atau jinak (benign).
Memahami alur pengambilan keputusan berdasarkan fitur morfologi sel dalam data.
Langkah-langkah Modelling#
Membagian Data (Train-Test Split)
Dataset dibagi menjadi data pelatihan dan data pengujian dengan proporsi 80:20 menggunakan fungsi train_test_split. Data pelatihan digunakan untuk membangun model, sementara data pengujian digunakan untuk mengukur performa model terhadap data yang belum pernah dilihat.Pembuatan dan Pelatihan Model
Model Decision Tree dibangun menggunakan pustaka sklearn.tree.DecisionTreeClassifier. Algoritma ini secara otomatis membentuk struktur pohon berdasarkan pembagian data yang memaksimalkan gain informasi (Information Gain) atau impuritas seperti Gini index.Prediksi dan Evaluasi Setelah model dilatih, dilakukan prediksi terhadap data pengujian. Evaluasi model dilakukan dengan metrik:
Akurasi: Tingkat ketepatan model dalam memprediksi label.
Confusion Matrix: Matriks yang menggambarkan jumlah prediksi benar dan salah.
Classification Report: Berisi metrik precision, recall, dan F1-score untuk tiap kelas.
from ucimlrepo import fetch_ucirepo
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier
from sklearn.metrics import classification_report, confusion_matrix, accuracy_score
# 1. Load data tanpa outlier
df = pd.read_csv('breast_cancer_tanpa_outlier.csv')
# 2. Pisahkan fitur dan target
X = df.drop(columns=['Diagnosis', 'ID', 'LOF Score', 'Prediksi'], errors='ignore')
y = df['Diagnosis']
# Split data: 80% training, 20% testing
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# Buat dan latih model Decision Tree
modeldt = DecisionTreeClassifier(random_state=42)
modeldt.fit(X_train, y_train)
# Prediksi data test
y_pred = modeldt.predict(X_test)
# Evaluasi model
print("Akurasi :", accuracy_score(y_test, y_pred))
print("\nConfusion Matrix:\n", confusion_matrix(y_test, y_pred))
print("\nClassification Report:\n", classification_report(y_test, y_pred))
Akurasi : 0.9795918367346939
Confusion Matrix:
[[65 1]
[ 1 31]]
Classification Report:
precision recall f1-score support
0 0.98 0.98 0.98 66
1 0.97 0.97 0.97 32
accuracy 0.98 98
macro avg 0.98 0.98 0.98 98
weighted avg 0.98 0.98 0.98 98
Evaluasi#
\( \begin{array}{lcccc} \hline \textbf{Model} & \textbf{Akurasi} \\ \hline \text{Gaussian Naive Bayes} & 0.9795918367346939 \\ \text{Decision tree} & 0.9795918367346939 \\ \hline \end{array} \)
Kedua model menunjukkan hasil akurasi yang sama, yaitu sebesar 97.96%, yang menunjukkan bahwa model mampu memprediksi jenis kanker dengan tingkat ketepatan yang sangat tinggi.
Hasil confusion matrix dari kedua model juga identik, menunjukkan bahwa:
Sebagian besar kasus jinak dan ganas berhasil diklasifikasikan dengan benar.
Jumlah kesalahan prediksi (false positive dan false negative) sangat sedikit, sehingga model dapat dikatakan cukup andal.
Deployment#
Menyimpan model#
import pickle
nama_model = "breast.pkl"
with open(nama_model, 'wb') as file:
pickle.dump(modeldt, file)
Hasil Web#
link web : https://pendata-bhcajlmnjr8buzvdjvu5am.streamlit.app/
